Views: 1038
di Salvatore Campo* e Angela Avenoso**

Per decenni i biologi hanno attribuito al DNA e alle proteine i ruoli più attivi nella cellula mentre all’RNA, sebbene indubbiamente importante, è stata attribuita una funzione di supporto. Oggi, invece, la visione, ormai semplicistica, del dogma centrale della biologia molecolare, che mette al centro del percorso dell’informazione genetica il DNA e attribuisce all’RNA un ruolo di intermediario passivo, è destinata sempre più ad essere messa in discussione.
La prima scoperta che ha ribaltato effettivamente il ruolo dell’RNA risale al 1986, quando il premio Nobel Thomas Cech ha dimostrato che esistono RNA, e non solo proteine, dotati di attività catalitica. La seconda grande rivoluzione è avvenuta nel 1993, grazie a uno studio sul verme Caenorhabditis elegans che ha messo in luce la presenza di piccoli RNA, chiamati lin4, in grado di bloccare la funzione di geni essenziali per lo sviluppo dell’animale.
Dopo 50 anni di ricerche centrate sul DNA, ci si è così accorti che oltre ai geni che codificano per proteine esiste un grandissimo numero di altri geni fondamentali per la cellula che non sono utili per la sintesi di proteine. Questi geni nel loro insieme vengono definiti non-coding RNA, o RNA non codificanti, un termine molto generico che racchiude diverse sottofamiglie con funzioni completamente diverse.
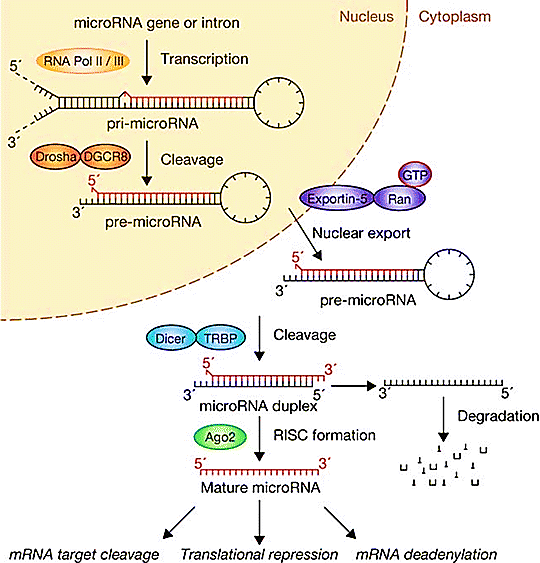
Una delle famiglie più studiate è quella dei microRNA (o miRNA), a cui appartiene proprio lin-4. Il nome è dovuto alle piccole dimensioni di questi RNA: 20-30 nucleotidi circa. La loro funzione è quella di regolare il metabolismo cellulare e la quantità di proteine prodotte: i miRNA sono perciò un sofisticato meccanismo di controllo dell’espressione genica. I micro-RNA finora identificati nell’Uomo superano i 5000 e molti di essi vengono espressi in modo tessuto-specifico. Sono codificati da particolari geni che, a loro volta, possono essere localizzati a livello di esoni, di introni (geni mirtroni) o di regioni intergeniche. Questi geni possono essere racchiusi in cluster o essere isolati e si possono trovare in forma monocistronica o policistronica. Questi geni vengono trascritti dalla RNA Polimerasi II e dalla RNA Polimerasi III in forma di lunghi RNA detti pri-miRNA o micro-RNA primari. In seguito ad un primo processo di maturazione nucleare, si forma una struttura intermedia più corta: il pre-micro-RNA o micro-RNA precursore. Attraverso uno specifico trasportatore, il pre-micro-RNA giunge nel citoplasma, ove subisce la definitiva maturazione da parte del microprocessore DICER. Il micro-RNA maturo rimane all’interno di DICER. Successivamente la proteina TRBP facilita l’ingresso del miRNA maturo associato a Dicer all’interno del complesso RISC, che contiene una delle 4 proteine catalitiche Argonauta. I due filamenti di RNA inseriti in RISC vengono quindi separati, analizzati e solo uno (filamento guida) farà parte del complesso microRISC attivo. L’azione del miRNA maturo sull’mRNA bersaglio dipende da un particolare appaiamento, detto appaiamento seed, il quale determinerà il blocco della traduzione. Il risultato sarà l’assenza del prodotto proteico finale. Di fatto, l’espressione dei microRNA risulta alterata in tutti i processi patologici, dall’Alzheimer ai disturbi cardiaci, ai tumori, e altri.
MicroRNA e tumori
Se i microRNA regolano l’espressione genica, è logico pensare che alcuni di essi controllino i livelli di tutti quei geni alterati coinvolti nei tumori. Alcuni miRNA, come i cinque appartenenti alla famiglia dei miR-200, alla famiglia 17-92 sono coinvolti in numerosissime neoplasie, ma esistono anche miRNA che svolgono un ruolo in ogni tipologia di cancro, evidenziando l’importanza sempre maggiore di queste molecole come biomarkers per la diagnosi e il controllo dell’evoluzione della patologia, ma anche come possibile strumento terapeutico.
I long non-coding RNA
Nell’ultimo decennio è stata scoperta un’altra classe di RNA non codificanti, i long non-coding RNA (long ncRNA). Sono lunghi più di 200 basi e sono ampiamente importanti nella stabilizzazione termodinamica delle proteine e nella loro regolazione genica post-traduzionale, controllando processi come la sintesi di proteine, la maturazione di RNA, il trasporto di una vasta gamma di molecole e i meccanismi di silenziamento genico. In quest’ultimo caso è stato osservato un loro attivo ruolo nel regolare la struttura della cromatina, interagendo sia con i complessi che modificano gli istoni sia con quelli che determinano la metilazione del DNA. Inoltre, sono in grado di interagire anche con complessi ribonucleoproteici regolatori della trascrizione, indirizzandoli in modo specifico sui relativi bersagli. In particolare, i lncRNA a localizzazione nucleare hanno il compito di modulare le modifiche epigenetiche rimodellando la cromatina, mentre nel citoplasma agiscono prevalentemente da regolatori post trascrizionali.
RNA Y
Gli RNA Y sono stati individuati per la prima volta in soggetti affetti da Lupus Eritematoso associati a specifiche proteine, Ro60 (RNA binding protein 60 o TROVE 2) e La (SSB small RNA binding exonuclease protection factor La). Si tratta di una classe di RNA non codificanti ampiamente espressi nel sistema nervoso centrale di tutti i vertebrati. L’uomo e tutti i primati possiedono 4 geni distinti in grado di codificare 4 differenti RNA Y (hY1, hY3, hY4 e hY5). La lunghezza degli RNA Y può variare tra 80 e 120 ribonucleotidi. Diversi studi hanno messo in relazione la loro dis-regolazione con differenti stati patologici, cancro incluso.
Anche se la trascrizione degli RNA Y è nota da diversi anni, i dati della letteratura in merito alle loro funzioni biologiche sono ancora pochi.
Gli RNA Y sono fondamentali per innescare il processo di replicazione del DNA.
L’RNA Y1 legato alla proteina Ro60 è in grado di assumere una struttura a toroide ellittico con un canale centrale in grado di permettere il passaggio di un filamento di RNA lineare. È stato accertato che, in seguito a processi in grado di alterare la struttura secondaria e/o terziaria degli RNA non codificanti (per es. radicali liberi, stati infiammatori, vari fattori di stress), similmente a quanto si osserva per le proteine, questi RNA misfolded (malpiegati) sono in grado di entrare attraverso il toroide, il quale, mediandone il refolding, (ripiegamento) permette a questi RNA di riprendere la loro funzione biologica. Altre funzioni note degli RNA Y sono sia favorire la terminazione di tutti i trascritti della RNA Polimerasi III (in particolare Y1 e Y3 sono in grado di legare la proteina nucleolina nel nucleolo) sia favorire l’assemblaggio di nuovi ribosomi.
Gli RNA Y sono anche ampiamente coinvolti negli stati infiammatori; in modo particolare è stato osservato che, durante il processo infiammatorio, i monociti/macrofagi incrementano notevolmente la loro espressione e, a loro volta, essi sono in grado di attivare il recettore TLR7 (Toll like receptor 7), di promuovere l’apoptosi caspasi 3 dipendente e modulare la risposta infiammatoria NF-kB dipendente.
Alla luce delle ricerche effettuate emerge chiaramente il ruolo degli RNA non codificanti in varie patologie. Ulteriori studi saranno necessari al fine di poterli utilizzare sia come markers sia come agenti o bersagli terapeutici alternative ai farmaci convenzionali.
*Professore Associato di Biologia Molecolare e **Referente InTIme, Dipartimento BIOMORF, Università di Messina